개발 로그 ④
개요
최근 다양한 산업 분야에서 딥러닝 모델이 활발하게 활용되고 있습니다. 이를 참고해 3D 의료 영상에 관한 작업을 진행했습니다. KiTS23 데이터셋을 활용하여 신장
및 종양 부위를 분할하는 모델을 구현하였습니다.
이 작업은 3D U-Net 아키텍처를 적용하여 구현했으며, 489개의 CT 영상을 8 : 1 : 1 비율로 학습, 검증, 테스트 데이터셋으로 분할해 학습 및
성능 평가를 진행했습니다.
사용 기술
데이터 수집 및 처리 과정
ㆍ 489개의 CT 영상으로 이뤄진 KiTS23 공개 데이터셋 사용
ㆍ 데이터 증강 → RandFlipd, RandAffined, Rand3DElasticd
ㆍ 데이터 분할 → Train (391개), Validation (49개), Test (49개)
모델 구조
ㆍ Downsampling
└ 4단계 :
[Double Conv → 1/2 × Downsample]
ㆍ Upsampling
└ 3단계 : [2
× Upsample → Skip Connection → Double Conv]
ㆍ Last Conv Layer
└ 최종 예측 결과
출력
학습 설정 및 결과
ㆍ Loss Function : DiceCELoss
ㆍ Optimizer : AdamW (learning rate = 0.0001, weight decay =
0.00001)
ㆍ Scheduler : ReduceLROnPlateau
ㆍ Epochs : 50회
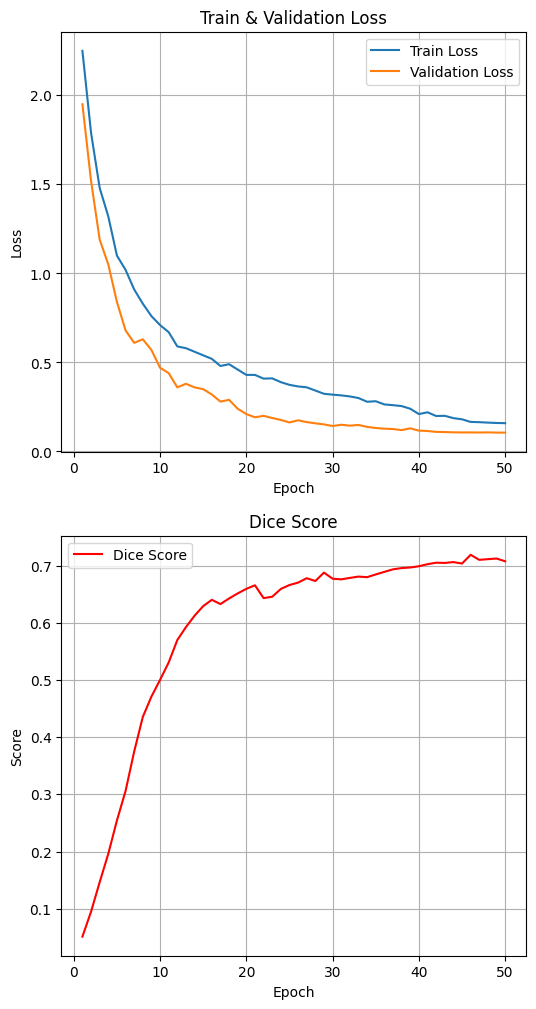
ㆍ Dice Score : 72.96%
ㆍ Training Curve :
고찰
ㆍ 초기 모델 구성
- 모델 최초
구현: 다운 샘플링시 최대 채널 수 512차원 까지 구현
- 결과 : 코랩 A100 환경에서 Out of
Memory 발생
ㆍ 개선 과정
- 다운 샘플링시
최대 채널 수를 256차원까지로 경량화
- 결과 : 50 에포크 이후에 Val Loss가
음수가 되는 현상 발생
ㆍ 최종 결과
- 50에포크
시점 모델을 최종 결과로 선정
- Dice Score :
72.96%
ㆍ 느낀 점
- Val
Loss의 음수값 → 원인 불명
└ 메모리 부족, 모델의 학습시간, 제한적인 PC
환경 등으로 충분한 실험이 어려움
└ ① 분할된 검증데이터에 종양 없는 데이터가 다수
포진되어 있을때 문제 발생 가능성
└ ② Batch size=2일 때, 한 Batch에
종양이 없는 데이터만 존재 시 문제 발생 가능성
└ ③ 학습이 진행되며 모델이 점점 확신을 가지면서
후반부 수치적 불안정성이 발생 했을 가능성