개발 로그 ②
개요
다양한 딥러닝 모델 경험, 비교를 위해 말라리아 세포감염 여부 분류 모델을 개발했으며, 데이터셋은 캐글의 Malaria Cell Images Dataset (원본
NIH)을 활용했습니다.
이 작업은 ResNet모델로 구현했으며, 총 약 27,500장의 이미지를 학습(80%), 테스트(20%) 데이터셋으로 분할하여 학습 및 성능 평가를
진행했습니다.
사용 기술
데이터 수집 및 처리 과정
ㆍ 총 27,558장의 공개 데이터셋 사용
ㆍ 데이터 증강 → Resize, HorizontalFlip, Rotation(20°), Normalize
ㆍ 데이터 분할 → Train (22,046장), Test (5,512장)
모델 구조
ㆍ 초기 Conv → BatchNorm → ReLU → MaxPool 구성;
└
Conv1 : 3
→ 64 (7 × 7, stride : 2)
ㆍ 4 × [BasicBlock → Residual connection 적용] 구성
└
Layer1 :
64 → 64
└
Layer2 :
64 → 128
└
Layer3 :
128 → 256
└
Layer4 :
256 → 512
ㆍ Adaptive Average Pooling → FC(512 → 1) → 최종 출력
학습 설정 및 결과
ㆍ Loss Function : BCEWithLogitsLoss
ㆍ Optimizer : SGD (learning rate = 0.1)
ㆍ Epochs : 20회
ㆍ Train Loss : 0.0993
ㆍ Training Curve :
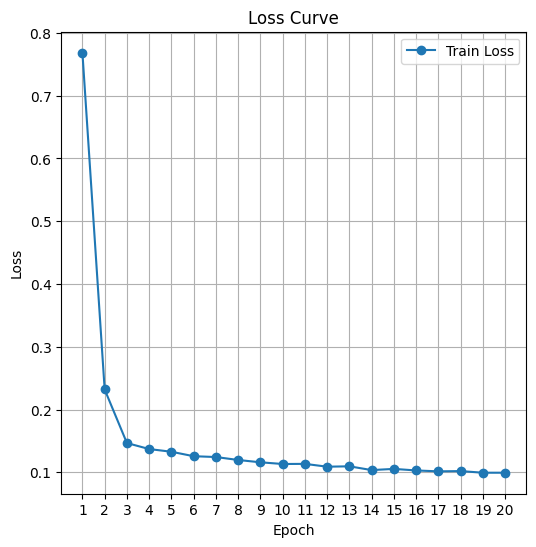
고찰
ㆍ 초기 데이터 전처리 및 모델 구성
- 데이터
증강에서 Resize 없었음
ㆍ 개선 과정
- 데이터로더의
입력 이미지 사이즈 일치 위해 Resize 추가
ㆍ 최종 결과
- 정확도 :
96.21%
ㆍ 느낀 점
- 일관된 입력
이미지 사이즈의 중요성
- Residual connection 구조가
gradient 소실 문제를 완화
└ 깊은 신경망에서도 안정적인 학습
가능